Μάθημα 1.1
Δημιουργία Ιστοσελίδας που περιέχει JSmol
Απεικόνιση και κίνηση μοριακού μοντέλου
Στόχος
Σε αυτό το μάθημα θα δημιουργήσουμε μια ιστοσελίδα με ένα αντικείμενο JSmol που απεικονίζει το μοριακό μοντέλο του νερού με μία απλή κίνηση.
Το τελικό αποτέλεσμα φαίνεται στο παρακάτω πλαίσιο:
Βήμα 1.
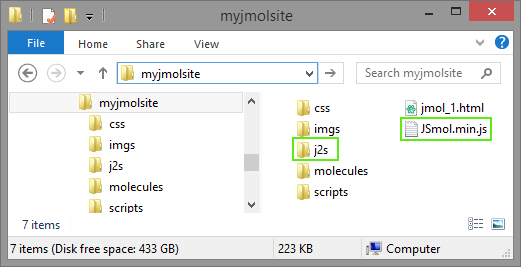
Δομή και Ιεραρχία Φακέλων και Αρχείων
- Δημιουργήστε ένα φάκελο του Ιστοχώρου και ονομάστε τον myjmolsite
-
Κατεβάστε στον υπολογιστή σας την τελευταία έκδοση του Jmol από το παρακάτω link:
https://sourceforge.net/projects/jmol/ -
Ανοίξτε ή αποσυμπιέστε το συμπιεσμένο αρχείο Jmol-14.xx.xx-binary.zip και στη συνέχεια ανοίξτε ή αποσυμπιέστε το συμπιεσμένο αρχείο jsmol.zip.
- Μέσα από τον φάκελο jsmol αντιγράψτε τον φάκελο j2s και το αρχείο JSmol.min.js και μεταφέρετέ τα στον φάκελο του ιστοχώρου σας, myjmolsite
-
Μέσα στον φάκελο myjmolsite δημιουργήστε τους παρακάτω κενούς φακέλους :
css, imgs, molecules και scripts -
Μέσα στον φάκελο myjmolsite δημιουργήστε ένα νέο αρχείο κειμένου (δεξί κλικ > New > Text Document ) και ονομάστε το jmol-1.html.
Βήμα 2.
Δημιουργία Ιστοσελίδας που περιέχει JSmol Object
- Ανοίξτε το αρχείο jmol-1.html με τον επεξεργαστή κειμένου Atom ή το Notepad++.
-
Δημιουργήστε μια κενή ιστοσελίδα (αντι)γράφοντας τον παρακάτω κώδικα στο αρχείο jmol-1.html:
jsmol-1.html
<!DOCTYPE html> <html lang="el" > <head> <meta charset="utf-8"> <title></title> </head> <body> </body> </html> -
Μέσα στο tag
head(αντι)γράψτε τον παρακάτω κώδικα:Ο νεοεισερχόμενος κώδικας εμφανίζεται με μαύρο τονισμένο background.jsmol-1.html<!DOCTYPE html> <html lang="el" > <head> <meta charset="utf-8"> <title></title> <script type="text/javascript" src="JSmol.min.js"> </script> <script> var jmolApplet0; var use = "HTML5" var s = document.location.search; Jmol.debugCode = (s.indexOf("debugcode") >= 0); jmol_isReady = function(applet) { } var JSmol_Applet = { width:"100%", height:"100%", debug: false, color: "#FFFFFF", use: "HTML5", j2sPath: "j2s", readyFunction: jmol_isReady, script: "", disableJ2SLoadMonitor: true, disableInitialConsole: true, allowJavaScript: true } </script> </head> <body> </body> </html>Στον παραπάνω κώδικα μπορείτε να προκαθορίσετε τις διαστάσεις (width, height) και το χρώμα (color) του Jmol.
Στο παραπάνω παράδειγμα οι διαστάσεις έχουν οριστεί σε ποσοστό 100% του γονικού tag και το χρώμα λευκό (#FFFFFF). Εναλακτικά μπορείτε να ορίσετε τις διαστάσεις σε pixels (px). -
Μέσα στο tag
bodyδημιουργήστε έναdiv. Oρίστε τις διαστάσεις του σε 400px και το περίγραμμα σε 1px με τη χρήση του attributestyle:jsmol-1.html<body> <div style="width:400px; height:400px; border:1px solid #333;" > </div> </body>Τοdivαυτό θα αποτελεί το γονικό tag ή 'περιτύλιγμα' (wrapper) μέσα στο οποίο θα ενθυλακωθεί η υλοποίηση του JSmol. Το JSmol θα πάρει τις διαστάσεις του περιτυλίγματος εφόσον στοheadέχουν προκαθοριστεί οι διαστάσεις του JSmol σε 100%. -
Μέσα στο
divαρχικοποιήστε το JSmol (αντι)γράφωντας τον παρακάτω κώδικαscript:jsmol-1.html<body> <div style="width:400px; height:400px; border:1px solid #333;" > <script> jmolApplet0 = Jmol.getApplet("jmolApplet0", JSmol_Applet); </script> </div> </body>Με την εντολήΤώρα το JSmol έχει εκκινηθεί και αν ανοίξετε το αρχείο jmol-1.html θα πρέπει να βλέπετε την παρακάτω εικόνα:Jmol.getApplet()αρχικοποιούμε μια υλοποίηση του JSmol, δηλαδή ένα JavaScript Object, και το αντιστοιχίζουμε σε μια μεταβλητή, στην περίπτωσή μας τηνjmolApplet0. Στη συνέχεια προκειμένου να επιδράσουμε στην συγκεκριμένη υλοποίηση του JSmol θα αναφερόμαστε στηνjmolApplet0. Με τον ίδιο τρόπο μπορούμε να δημιουργήσουμε πολλαπλές υλοποιήσεις του JSmol, π.χ.jmolApplet1 ,jmolApplet2, jmolApplet3κτλ.Προσοχή! Το JSmol σε τοπικό υπολογιστή τρέχει μόνο στο Firefox, ενώ διαδικτυακά τρέχει σε όλους τους browsers και σε όλα τα λειτουργικά. Για αυτό κατα την ανάπτυξη της ιστοσελίδας πρέπει να χρησιμοποιείτε το Firefox.
Βήμα 3.
Εισαγωγή μοριακού μοντέλου στο JSmol
Τέτοια αρχεία μπορείτε να δημιουργήσετε σε λογισμικά μοριακής οπτικοποίησης όπως τo Avogadro το οποίο είναι ελέυθερο https://avogadro.cc/, το Chem3D και GaussView.
Ενναλακτικά μπορείτε να κατεβάσετε αρχεία μοριακών μοντέλων από χημικές βάσεις δεδομένων οπως η Pubchem, η Chemspider, η NIST και η RCSB PDB.
-
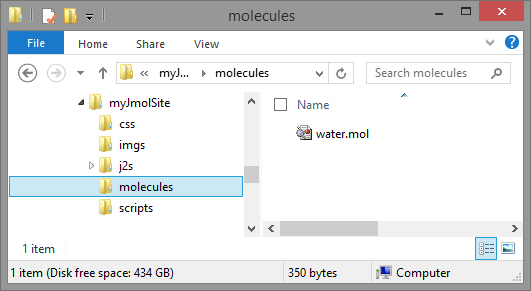
Δημιουργήστε ένα αρχείο .mol με την γεωμετρία του μορίου του νερού, ονομάστε το water.mol και τοποθετήστε το μέσα στον φάκελο molecules.
-
Μέσα στο tag
scriptπου βρίσκεται στο tagdivτουbody, (αντι)γράψτε τον παρακάτω κώδικα:jsmol-1.html<div style="width:400px; height:400px; border:1px solid #333; " > <script> jmolApplet0 = Jmol.getApplet("jmolApplet0", JSmol_Applet); Jmol.script(jmolApplet0, "load molecules/water.mol;"); </script> </div>H εντολή (function)
Jmol.script()επιδρά σε μια υλοποίηση (object) JSmol και εκτελεί κώδικα της scripting γλωσσας του JSmol.Δέχεται δύο παραμέτρους: πρώτα ορίζεται το object του JSmol στο οποίο επιδρά (
jmolApplet0) και στη συνέχεια μέσα σε εισαγωγικά γράφεται ο κώδικας της scripting γλώσσας του JSmol, π.χ."load molecules/water.mol;".Οι εντολές (functions) του Jmol JavaScript Object περιγράφονται στην ιστοσελίδα: http://wiki.jmol.org/index.php/Jmol_JavaScript_Object/Functions
Το πλήρες εγχειρίδιο της scripting γλώσσας του JSmol βρίσκεται στην ιστοσελίδα: https://chemapps.stolaf.edu/jmol/docs/
- Ανανεώστε την ιστοσελίδα jmol-1.html και δείτε το μοριακό μοντέλο του νερού. Περιστρέψτε το μοντέλο με το ποντίκι και ζουμάρετε με την ροδέλα του κύλισης. Το αποτέλεσμα εμφανίζεται παρακάτω:
Βήμα 4.
Βελτιστοποίηση απεικόνισης του JSmol
set antialiasDisplay on
-
(Αντι)γράψτε την εντολή
set antialiasDisplay on;μέσα στα εισαγωγικά του δεύτερου μέρους της εντολήςJmol.script(), μετά την εντολήload...jsmol-1.html<script> jmolApplet0 = Jmol.getApplet("jmolApplet0", JSmol_Applet); Jmol.script(jmolApplet0, "load molecules/water.mol; set antialiasDisplay on;"); </script>Στο δεύτερο μέρος της Javascript εντολήςJmol.script()μπορούν να εισαγχούν διαδοχικά πολυάριθμες εντολές της scripting γλωσσας του Jmol. Προσοχή! Το τέλος κάθε scripting εντολής πρέπει να δηλωνεται με το ελληνικό ερωτηματικό; - Ανανεώστε την ιστοσελίδα και παρατηρείστε την βελτίωση της απεικόνισης. Το αποτέλεσμα φαίνεται παρακάτω:
Βήμα 5.
Καθορισμός προσανατολισμού μοριακού μοντέλου
Μπορείτε να καθορίσετε τον προσνατολισμό και γενικότερα τη θέαση του μοριακού μοντέλου με την scripting εντολή moveto. Με αυτή την εντολή μπορείτε επίσης να δημιουργήσετε animations.
Η εντολή moveto περιέχει μια πληθώρα τιμών που περιγράφουν τη θέση, την περιστροφή και τη μετατόπιση του μονέλου, τον ορισμό και την περιστροφή των καρτεσιανών αξόνων, το κέντρο και την ακτινα περιστροφής του μοντέλου, το ζουμ, τη θέση και την περιστροφή της καμερας.
Θα δούμε παρακάτω οτι μπορούμε πολύ εύκολα να καταγράφουμε και να εφαρμόζουμε αυτές τις τιμές χρησιμοποιώντας την κονσόλα του JSmol.
- Προσανατολίστε το μόριο στην επιθυμητό προσανατολισμό.
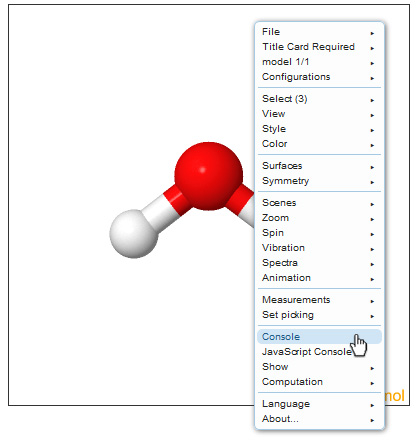
- Κάντε δεξι κλικ μέσα στο JSmol.
-
Στο αναδυόμενο μενού που εμφανίζεται επιλέξτε Console.
-
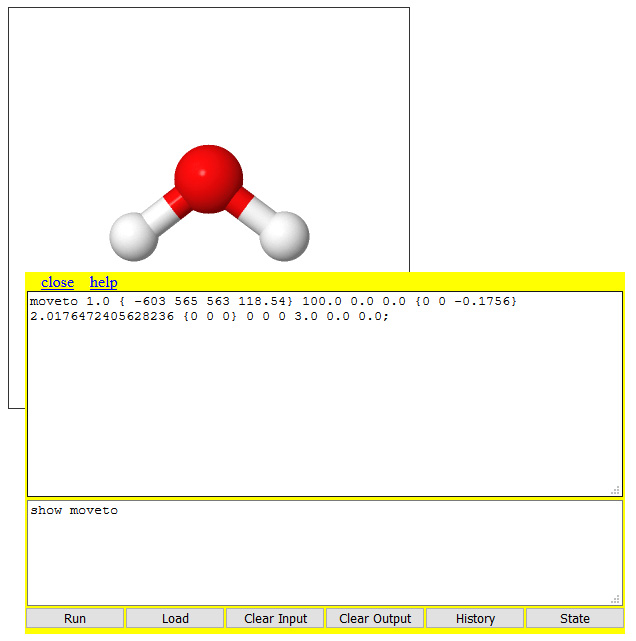
Γράψτε στο κάτω μέρος της κονσόλας που άνοιξε την εντολή
show movetoκαι πατήστε Enter.
Παρατηρείστε στο πάνω μέρος της κονσόλας την εντολήmovetoμε όλες τις τιμές των παραμέτρων που καθoρίζουν την τρέχουσα θέαση και προσανατολισμό του μορίου.
 Από όλες τις τιμές της εντολής
Από όλες τις τιμές της εντολήςmovetoμας ενδιαφέρει μόνο ο πρώτος αριθμός, ο οποίος εκφράζει τον χρόνο σε δευτερόλεπτα που απαιτείται για να μεταβεί το μοριακό μοντέλο από τον τρέχοντα προσανατολισμό στον τελικό προσανατολισμό, δημιουργώντας μια ομαλή κίνηση. -
Αντιγράψτε από την κονσόλα την εντολή
movetoμε όλους τους αριθμούς και επικολήστε την στo δεύτερο μέρος της εντολήςJmol.script()μετά την εντολήset antialiasDisplay on;jsmol-1.html<script> jmolApplet0 = Jmol.getApplet("jmolApplet0", JSmol_Applet); Jmol.script(jmolApplet0, "load molecules/water.mol; set antialiasDisplay on; moveto 1.0 { -603 565 563 118.54} 100.0 0.0 0.0 {0 0 -0.1756} 2.0176472405628236 {0 0 0} 0 0 0 3.0 0.0 0.0;"); </script>Η εντολές της scripting γλωσσας του JSmol πρέπει να γράφονται διαδοχικά σε μία γραμμή (χωρίς return ή enter), και να καταλήγουν σε ελληνικό ερωτηματικό.Παρατηρείστε στο παρακάτω πλαίσιο το αποτέλεσμα της εντολήςmoveto. Πατήστε το κουμπί Ανανέωση για να ξαναφορτώσετε το πλαίσιο.Κίνηση του μορίου διαρκει 1 sec γιατί τόσο ορίστηκε στον πρώτο αριθμό της εντολήςmoveto. Ορίστε αυτήν την τιμή0.0και το μοντέλο θα απεικονίζεται κατευθείαν στον τελικό προσανατολισμό. Παρατηρείστε το αποτέλεσμα πατώντας Ανανέωση στο παρακάτω πλαίσιο.jsmol-1.html<script> jmolApplet0 = Jmol.getApplet("jmolApplet0", JSmol_Applet); Jmol.script(jmolApplet0, "load molecules/water.mol; set antialiasDisplay on; moveto 0.0 { -603 565 563 118.54} 100.0 0.0 0.0 {0 0 -0.1756} 2.0176472405628236 {0 0 0} 0 0 0 3.0 0.0 0.0;"); </script>
Βήμα 6.
Δημιουργία κίνησης μοριακού μοντέλου
moveto μπορείτε να δημιουργήσετε κίνηση (animation) του μοριακού μοντέλου από έναν αρχικό προσανατολισμό σε έναν τελικό προσανατολίσμό.
- Περιστέψτε το μοριακό μοντέλο σε τυχαίο προσανατολισμό και μειώστε το ζουμ έτσι ώστε το μοριακό μονέλο να είναι πολύ μικρό.
- Ανοίξτε την κονσόλα: δεξί κλικ > Console.
- Γράψτε στο κάτω μερός της κονσόλας την εντολή
show movetoκαι πατήστε enter - Αντιγράψτε όλο το
movetoκαι επικολήστε το πρίν το υπάρχωνmovetoμέσα στο δεύτερο μέρος της εντολήςJmol.script() -
Ορίστε τον χρόνο, δηλ. τον πρώτο αριθμό, του πρώτου
movetoσε0.0και τον χρόνο του δεύτερουmovetoσε1.0jsmol-1.html<script> jmolApplet0 = Jmol.getApplet("jmolApplet0", JSmol_Applet); Jmol.script(jmolApplet0, "load molecules/water.mol; set antialiasDisplay on; moveto 0.0 { 671 -140 728 153.39} 5.0 0.0 0.0 {0 0 -0.1756} 2.0176472405628236 {0 0 0} 0 0 0 3.0 0.0 0.0; moveto 1.0{ -603 565 563 118.54} 100.0 0.0 0.0 {0 0 -0.1756} 2.0176472405628236 {0 0 0} 0 0 0 3.0 0.0 0.0;"); </script>Τώρα το μοριακό μοντέλο εμφανίζεται με ομαλή κίνηση από το βάθος προς τον τελικό προσανατολισμό.
Η εισαγωγή κώδικα scripting Jmol στο δεύτερο μέρος της JavaScript εντολής Jmol.script() γίνεται δύσχρηστη καθώς όλες οι εντολές πρέπει να γράφονται σε μια σειρά.
Η scripting γλώσσα του Jmol έχει πολλές δυνατότητες και μας επιτρέπει να αναπτύσουμε πολύπλοκα σενάρια μοριακής οπτικοποίησης γράφοντας τον κατάλληλο κώδικα.
Θα δούμε στη συνέχεια ότι ο κώδικας scripting Jmol είναι καλύτερα να γράφεται σε εξωτερικά αρχεία κειμένου με κατάληξη .spt
Λήψεις
Downloads
- Αρχείο γεωμετρίας του νερού: water.mol
- Φάκελος ιστοχώρου με κενή ιστοσελίδα JSmol: Jsmol-starter-site.zip
