Μάθημα 2.4
Οπτικοποίηση Μοριακών Τροχιακών
Στόχος
Σε αυτό το μάθημα θα δημιουργήσουμε μια ιστοσελίδα με ένα αντικείμενο JSmol που απεικονίζει τα μοριακά τροχιακά του μορίου του νερού.
Οι πληροφορίες για τη δημιουργία των ισοεπιφανειών των τροχιακών εξάγονται από το αρχείο εξόδου ενός κατάλληλου κβαντοχημικού υπολογισμού του λογισμικού Gaussian.
Το τελικό αποτέλεσμα φαίνεται στο παρακάτω πλαίσιο:
Βήμα 1.
Εκτέλεση κατάλληλου Κβαντοχημικού Υπολογισμού για το JSmol
Το JSmol μπορεί να διαβάζει και να οπτικοποιεί τα αποτελέσματα κβαντοχημικών υπολογισμών του πακέτου Gaussian.
Για την οπτικοποίηση των μοριακών τροχιακών πρέπει να εισαχθούν τα keywords Pop = Full και gfprint στο αρχείο υπολογισμού του Gaussian .gjf.
Εκτελέστε τον υπολογισμό ακολουθώντας τα παρακάτω βήματα.
Εναλακτικά, μπορείτε να κατεβάσετε τα αρχεία εισόδου και εξόδου του υπολογισμού Gaussian από εδώ:
water.gjf WATER.LOG
Pop = Full και gfprint .-
Δημιουργήστε ένα αρχείο κειμένου μέσα στον φάκελο molecules και ονομάστε το water.gjf

-
Ανοίξτε το αρχείο water.gjf με Notepad++ και αντιγράψτε το παρακάτω κείμενο:
water.gjf
# opt b3lyp/6-31g gfprint pop=full water for JSmol 0 1 O 0.000 0.000 0.000 H 0.785 -0.427 0.000 H -0.785 -0.427 0.000
Μπορείτε να κατεβάσετε το αρχείο εδώ: water.gjf -
Εκτελέστε τον υπολογισμό με το Gaussian.
Το αποτέλεσμα του υπολογισμού είναι ένα αρχείο με κατάληξη .log ή .out

Βήμα 2.
Δημιουργία αρχικής ιστοσελίδας με οπτικοποίηση μορίου από υπολογισμό
-
Δημιουργήστε μια ιστοσελίδα με ένα αντικείμενο JSmol και ονομάστε την jsmol-2-4.html
Ο κώδικας της ιστοσελίδας φαίνεται παρακάτω:
jsmol-2-4.html<!DOCTYPE html> <html lang="el" > <head> <meta charset="utf-8"> <title></title> <script type="text/javascript" src="JSmol.min.js"> </script> <script> var jmolApplet0; var use = "HTML5" var s = document.location.search; Jmol.debugCode = (s.indexOf("debugcode") >= 0); jmol_isReady = function(applet) { } var JSmol_Applet = { width:"100%", height:"100%", debug: false, color: "#FFFFFF", use: "HTML5", j2sPath: "j2s", readyFunction: jmol_isReady, script: "", disableJ2SLoadMonitor: true, disableInitialConsole: true, allowJavaScript: true } </script> </head> <body> <div style="width:450px; height:400px; border:1px solid #333;" > <script> jmolApplet0 = Jmol.getApplet("jmolApplet0", JSmol_Applet); </script> </div> </body> </html> -
Παρατηρείστε το αποτέλεσμα στο παρακάτω πλαίσιο.
Το JSmol διαβάζει όλες τις γεωμετρίες του μορίου που βρίσκονται στο αρχείο εξόδου του Gaussian και μπορεί να οπτικοποιήσει κάθε μια από αυτές.
Κάθε γεωμετρία αντιστοιχεί σε ένα μοντέλο (model) του JSmol
Σε έναν υπολογισμό βελτιστοποίησης γεωμετρίας υπάρχουν δύο models για κάθε βήμα (input orientation και standard orientation).
To JSmol αρχικά οπτικοποιεί το πρώτο model του αρχείου, ενώ το ζητούμενο είναι η οπτικοποίηση του τελευταίου model.
Μπορούμε να δούμε τα models που υπάρχουν σε ένα JSmol με δεξί κλικ > model, όπως φαίνεται παρακάτω:
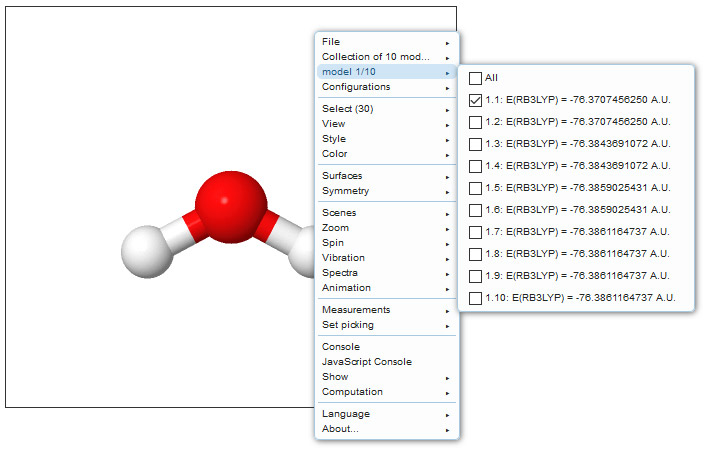 Η επιλογή του model που απεικονίζεται στο JSmol γίνεται με την εντολή
Η επιλογή του model που απεικονίζεται στο JSmol γίνεται με την εντολή
frame x.x, όπου x.x είναι ο αριθμός (id) του model. -
Φορτώστε το τελευταίο model του αρχείου εισάγοντας την εντολή frame 1.10 μετά την εντολή load:
jsmol-2-4.html
<script> jmolApplet0 = Jmol.getApplet("jmolApplet0", JSmol_Applet); Jmol.script(jmolApplet0, "set antialiasDisplay on; load molecules/WATER.LOG; frame 1.10;") </script>Παρατηρείστε το αποτέλεσμα στο παρακάτω πλαίσιο. Κάντε δεξί κλικ και παρατηρείστε το επιλεγμένο μοντέλο.
-
Προσανατολίσουμε το μόριο στο επίπεδο της οθόνης εκτελώντας διαδοχικά τις εντολές rotate z 90; rotate x -90; και zoom 70; μετά την εντολή frame 1.10
jsmol-2-4.html<script> jmolApplet0 = Jmol.getApplet("jmolApplet0", JSmol_Applet); Jmol.script(jmolApplet0, "set antialiasDisplay on; load molecules/WATER.LOG; frame 1.10; rotate z 90; rotate x -90; zoom 70;") </script>Παρατηρείστε το αποτέλεσμα στο παρακάτω πλαίσιο.
Βήμα 3.
Οπτικοποίηση Μοριακών Τροχιακών
Το JSmol μπορεί να οπτικοποιήσει τα Μοριακά Τροχιακά (ΜΟ) από το αρχείο εξόδου του Gaussian.
Για να δείτε τα MOs του μορίου κάντε δεξί κλικ > surfaces > Molecular Orbitals, όπως φαίνεται παρακάτω:
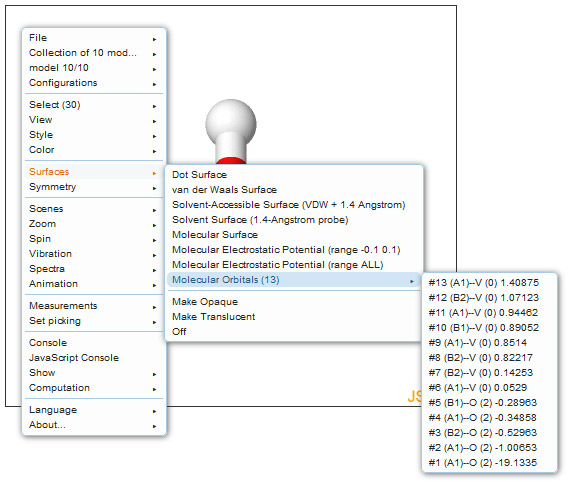
Η οπτικοποίηση των ΜΟ γίνεται με την εντολή mo x , όπου x είναι ο αριθμός του τροχιακού.
Επίσης, η εντολή μπορεί να έχει τη μορφή mo homo±x ή mo lumo±x όπου x είναι ακέραιος αριθμός.
Πειραματίσου με την οπτικοποίηση τροχιακών στο παρακάτω παράδειγμα εισάγοντας τον αριθμό του τροχιακού και πατώντας run.
Προσοχή!
Η οπτικοποίηση των ΜΟ γίνεται μόνο για το τελευταίο model που υπάρχει στο αρχείο εξόδου του υπολογισμού.
Στη συνέχεια θα δημιουργήσουμε ένα αναδυόμενο μενού επιλογής των κατειλημμένων μοριακών τροχιακών σθένους και του LUMO.
- Εισάγετε τον παρακάτω κώδικα για τη δημιουργία αναδυόμενου μενού:
jsmol-2-4.html
<script> jmolApplet0 = Jmol.getApplet("jmolApplet0", JSmol_Applet); Jmol.script(jmolApplet0, "set antialiasDisplay on; load molecules/WATER.LOG; frame 1.10; rotate z 90; rotate x -90; zoom 70;") Jmol.jmolMenu(jmolApplet0, [ [ "mo off; ", "Επίλεξε Μοριακό Τροχιακό "], [ "mo lumo; ", "LUMO "], [ "mo homo; ", "HOMO "], [ "mo homo-1; ", "HOMO-1 "], [ "mo homo-2; ", "HOMO-2 "], [ "mo homo-3; ", "HOMO-3 "] ]); </script> - Παρατηρείστε το αποτέλεσμα στο παρακάτω πλαίσιο.
Βήμα 4.
Βελτιστοποίηση Απεικόνισης Μοριακών Τροχιακών
Παρατηρούμε ότι η οπτικοποίηση των ΜΟ γίνεται αρχικά με μορφή πλέγματος (mesh) καθώς και ότι εμφανίζονται αυτόματα πληροφορίες για το απεικονιζόμενο τροχιακό.
Μπορούμε να βελτιστοποιήσουμε την απεικόνιση των τροχιακών δημιουργώντας ημιδιαφανείς ισοεπιφάνειες και αποκρύπτοντας το κείμενο.
- H απόκρυψη του πλέγματος γίνεται με την εντολή mo nomesh;
- Η απεικόνιση του τροχιακού ως ισοεπιφάνεια γίνεται με την εντολή mo fill;
- Η διαφάνεια της ισοεπιφάνειαs ορίζεται με την εντολή mo translucent x.x; , όπου x.x είναι δεκαδικός αριθμός που παίρνει τιμές από 0.0 (καμία διαφάνεια) έως 0.99 (πλήρης διαφάνεια)
- Εισάγετε τις εντολές mo nomesh; mo fill; mo translucent 0.3; μετά την εντολή load όπως φαίνεται παρακάτω:
jsmol-2-4.html
<script> jmolApplet0 = Jmol.getApplet("jmolApplet0", JSmol_Applet); Jmol.script(jmolApplet0, "set antialiasDisplay on; load molecules/WATER.LOG; frame 1.10; rotate z 90; rotate x -90; zoom 70; mo nomesh; mo fill; mo translucent 0.3;") Jmol.jmolMenu(jmolApplet0, [ [ "mo off; ", "Επίλεξε Μοριακό Τροχιακό "], [ "mo lumo; ", "LUMO "], [ "mo homo; ", "HOMO "], [ "mo homo-1; ", "HOMO-1 "], [ "mo homo-2; ", "HOMO-2 "], [ "mo homo-3; ", "HOMO-3 "] ]); </script> - Παρατηρείστε το αποτέλεσμα στο παρακάτω πλαίσιο.
- Η απόκρυψη του κειμένου πληροφοριών του τροχιακού γίνεται με την εντολή mo titleformat ''Εισάγετε την εντολή mo titleformat ''; μετά την εντολή load όπως φαίνεται παρακάτω:jsmol-2-4.html
<script> jmolApplet0 = Jmol.getApplet("jmolApplet0", JSmol_Applet); Jmol.script(jmolApplet0, "set antialiasDisplay on; load molecules/WATER.LOG; frame 1.10; rotate z 90; rotate x -90; zoom 70; mo nomesh; mo fill; mo translucent 0.3; mo titleformat '';") Jmol.jmolMenu(jmolApplet0, [ [ "mo off; ", "Επίλεξε Μοριακό Τροχιακό "], [ "mo lumo; ", "LUMO "], [ "mo homo; ", "HOMO "], [ "mo homo-1; ", "HOMO-1 "], [ "mo homo-2; ", "HOMO-2 "], [ "mo homo-3; ", "HOMO-3 "] ]); </script> - Παρατηρείστε το αποτέλεσμα στο παρακάτω πλαίσιο.
Λήψεις
Downloads
- Αρχείο εξόδου υπολογισμού βελτιστοποίησης του νερού: WATER.LOG
- Φάκελος ιστοχώρου με κενή ιστοσελίδα JSmol: Jsmol-starter-site.zip
- Φάκελος ιστοχώρου τελικού αποτελέσματος του μαθήματος: lesson24-full-site.zip
